近日,我院植物免疫团队在Plant Communications期刊在线发表了题为“Development and application of the GenoBaits®WheatSNP16K array to accelerate wheat genetic research and breeding”的研究论文。我院博士后刘胜杰为论文第一作者,曾庆东副研究员、康振生教授为论文通讯作者。农学院在读博士生相明杰和王晓婷为论文共同第一作者,韩德俊教授为共同通讯作者。
单核苷酸多态性(SNP)标记被广泛用于小麦种质资源挖掘、基因/QTL的鉴定,重要性状解析等遗传研究以及分子育种。从头组装、全基因组重测序等方案是最全面的鉴定大量种质资源中DNA变异的方法,但是由于小麦基因组庞大,采用该方案价格太过昂贵。SNP芯片技术是一种性价比相对较高的替代方案,广受研究人员和育种家的喜爱。然而,当前小麦中已有的SNP芯片均采用固相芯片技术,往往需要特定的测序平台,并且只能检测固定的SNP位点,基因分型成本较高,需要新的SNP基因分型平台。
因此,该研究利用课题组1520份小麦660K SNP芯片数据和20份小麦重测序数据产生的变异数据集,开发了基于靶向测序和液相捕获技术的GenoBaits®WheatSNP16K(GBW16K)液相芯片,并将其成功应用于小麦种质资源的多样性评估和亲缘关系分析。此外,利用GBW16K芯片该研究构建了由CIMMYT小麦品系Yaco'S与中国地方品种铭贤169的RIL群体遗传连锁图谱,并通过全基因组连锁分析鉴定出4个成株期抗条锈病位点,其中2BS、2BL和3BS位点来自抗病亲本Yaco“S”,7DS位点来自铭贤169。根据精细定位、分子标记以及相对位置分析,2BS、3BS、7DS位点分别为 Yr27 、 Yr30 和 Yr18 基因,2BL位点为新抗病位点不同于以往报道的基因/QTL。在1520份小麦中,QYr.nwafu-2BL.4区段存在3种主要的单倍型,其中Hap_1(Yaco“S”)为优异单倍型,具有更低的病害反应型和严重度。综合共线性分析、基因注释、基因表达和SNP数据,共筛选到6个注释基因作为QYr.nwafu-2BL.4的候选基因。
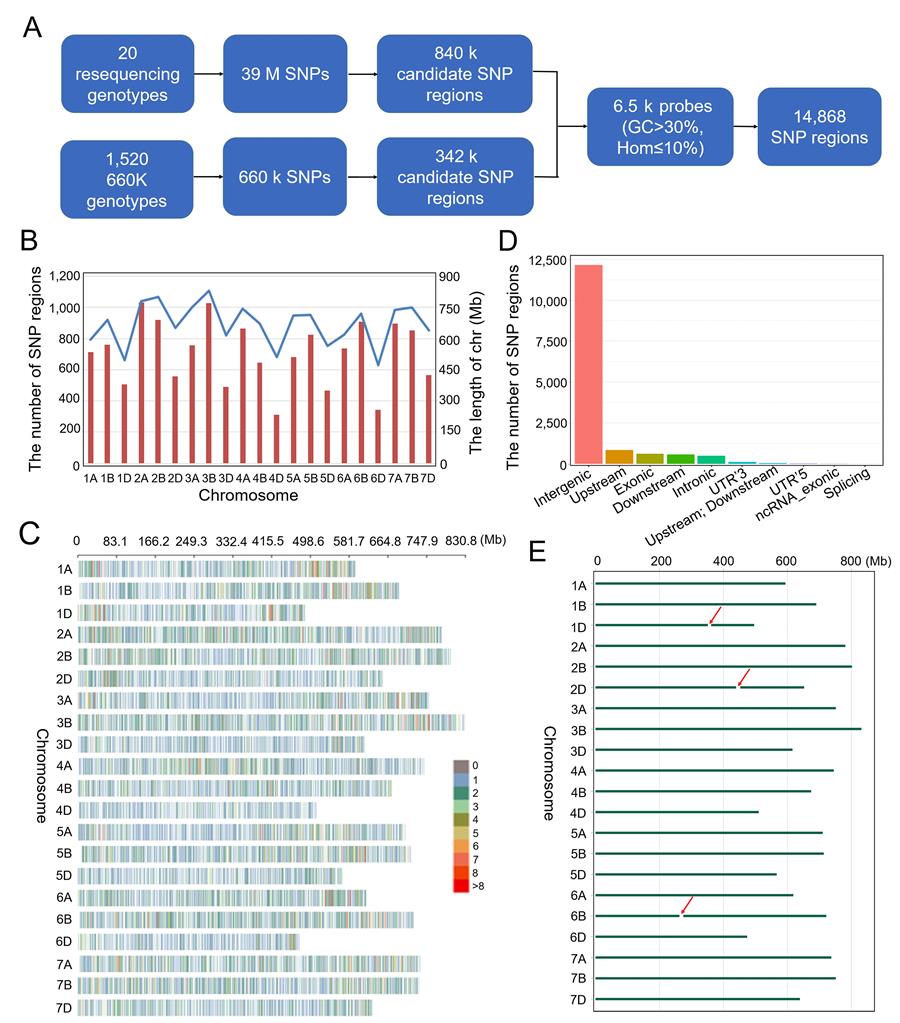
GBW16K液相芯片的设计与统计信息
该研究开发的GBW16K液相芯片,相较小麦中已有的15K、35K、50K、55K、90K、660K和820K等固相SNP芯片具有技术要求更低、经济成本更低和检测位点更灵活等优势,将加速小麦遗传研究和分子育种改良,定位的主效抗病位点 QYr.nwafu-2BL.4 为小麦遗传改良提供了新的基因资源。
农学院郑炜君副教授,李春莲副教授和吴建辉副教授也参与了该研究,国际玉米小麦改良中心Ravi P. Singh和Sridhar Bhavani教授为该研究提供了帮助。作物抗逆与高效生产全国重点实验室实验平台提供了技术支持。该研究得到了国家重点研发计划、国家自然科学基金项目等项目的资助。
原文链接:https://doi.org/10.1016/j.xplc.2024.101138
编辑:刘小凤
审核:郭 军